 同社は2001年6月設立。受託開発をメインとする独立系ソフトウエア会社で、バイオインフォマティクスやヘルスケア領域のシステム開発からスタートし、現在では組み込み系、通信系、ERP(エンタープライズリソースプランニング)系のビジネスも手がけている。沖縄と東京の2拠点での開発のほか、スリランカでのオフショア開発にも対応できることが強みである。
同社は2001年6月設立。受託開発をメインとする独立系ソフトウエア会社で、バイオインフォマティクスやヘルスケア領域のシステム開発からスタートし、現在では組み込み系、通信系、ERP(エンタープライズリソースプランニング)系のビジネスも手がけている。沖縄と東京の2拠点での開発のほか、スリランカでのオフショア開発にも対応できることが強みである。
アクシオヘリックスがゲノム研究支援ソフトを外販
理化学研究所のOmicBrowseをカスタマイズ、公開/内部データを統合解析
2008.05.31−アクシオヘリックス(本社・沖縄県那覇市、シバスンタラン・スハルナン社長)は、生命科学研究に役立つプラットホームソフトウエア「OmicBrowse」(オミックブラウズ)の販売を開始した。もともとは理化学研究所が開発したものだが、今回、外部に対してカスタマイズと再販売が行える権利をアクシオヘリックスが取得した。内部の研究データを、ゲノム関連の公開データベースと合わせて統合的に解析したい製薬・バイオ企業や大学向けに提供していく。ライセンス価格は98万円から。
 同社は2001年6月設立。受託開発をメインとする独立系ソフトウエア会社で、バイオインフォマティクスやヘルスケア領域のシステム開発からスタートし、現在では組み込み系、通信系、ERP(エンタープライズリソースプランニング)系のビジネスも手がけている。沖縄と東京の2拠点での開発のほか、スリランカでのオフショア開発にも対応できることが強みである。
同社は2001年6月設立。受託開発をメインとする独立系ソフトウエア会社で、バイオインフォマティクスやヘルスケア領域のシステム開発からスタートし、現在では組み込み系、通信系、ERP(エンタープライズリソースプランニング)系のビジネスも手がけている。沖縄と東京の2拠点での開発のほか、スリランカでのオフショア開発にも対応できることが強みである。
今回のOmicBrowseは、理研総合データベース(http://omicspace.riken.jp/db/index.html.ja)のゲノムデータベース検索システム(http://omicspace.riken.jp/db/genome.html.ja)として誰でも実際に使用することができるほか、2006年11月からはオープンソースとしてプログラム自体が無償公開されている。今回は、理研からバイオインフォマティクス領域の実績と技術力を評価され、これをカスタマイズして販売できる権利を、理研から取得したかたちになる。
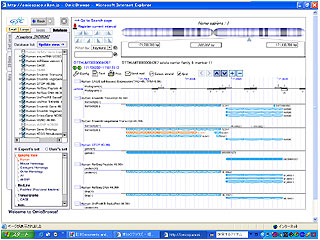
OmicBrowseは、ゲノム上に存在する遺伝子などのさまざまなアノテーション情報に関する多数のデータベース群を同時に検索して、その結果を可視化する機能を持つ。データサーバーとビューアーから構成されており、データサーバーは強力なデータ統合機能と豊富な拡張性を持ち、ビューアーは表示範囲に限界がないという特徴がある。
従来のソフトは、アノテーションを単純に表示するものがほとんどで、データ量が多い場合には表示に限界があった。これに対し、OmicBrowseでは表示範囲に制限がなく、データが多い場合には統計的な指標を瞬時に計算して表示することができる。そこからキーワードで重要な遺伝子を絞り込むことにより、病気の原因遺伝子を探索するなどの作業が大幅に効率化されるという。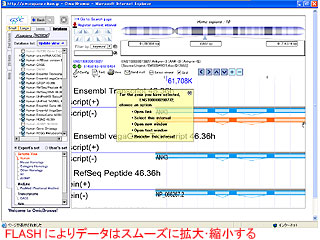
また、ゲノム統合データベースは数百から千種類のデータベースを包含することになるが、従来のソフトがそれぞれのデータベースにアクセスするのに対し、OmicBrowseではまとめて1回のアクセスで済むため、ネットワーク負荷が大幅に軽減される。データサーバーを複数台連結して、大量のデータの分散共有を図ることができることから、内部データ量の増加にも柔軟に対応することが可能だ。
ビューアーは、マクロメディアのFLASH技術を使って構築されており、通常のウェブブラウザーを用いて高品位な表示が行える。とくに、アノテーションをベクトルイメージで表示するため、データが多くて画像が画面をはみ出すということもなく、高画質のまま滑らかに拡大縮小を行うことができる。全体像の俯瞰から注目部分の拡大まで自由自在に画面を操作することが可能。
OmicBrowseは基本的にはオープンソースとして入手できるものの、公開データと内部データを比較検討できるようにしたり、理研サイトに載っていない公開データを登録したり、関連機能を追加したりするカスタマイズをユーザー自身が行うことは難しい。そこで、そもそもの開発元としてノウハウを持つ同社が、コンサルティングを含めて個別のユーザーニーズに対応し、カスタマイズを施して提供していくことにした。