 MOEは、計算化学や分子モデリング機能のほか、大量の化合物データを効率良く処理するケムインフォマティクス機能、たん白質の立体構造予測などのバイオインフォマティクス機能などを兼ね備えた統合ソフト。独自のプログラミング言語である“SVL”を中心にした開発環境が統合されており、アプリケーションを開発したりカスタマイズしたり、オリジナルのメニューやボタンバーを追加したりすることが簡単に行える。
MOEは、計算化学や分子モデリング機能のほか、大量の化合物データを効率良く処理するケムインフォマティクス機能、たん白質の立体構造予測などのバイオインフォマティクス機能などを兼ね備えた統合ソフト。独自のプログラミング言語である“SVL”を中心にした開発環境が統合されており、アプリケーションを開発したりカスタマイズしたり、オリジナルのメニューやボタンバーを追加したりすることが簡単に行える。
菱化システムがMOE向けの3D-QSARツールを開発
重ね合わせ・最良モデル選択など自動化、無償で提供開始
2010.07.18−菱化システムは、加CCGが開発した分子設計統合システム「MOE」に3D-QSAR機能を追加する新モジュール「MOE-AutoGPA」を独自に開発し、MOEユーザー向けに無償で公開開始した。自動的にさまざまな配座を使用した3D-QSARモデルを構築し、最良なモデルを簡単に選び出すことができる。スクリーニングや分子設計につなげることも可能。6月から7月にかけて行われた北米および日本のユーザー会で紹介され、早くも関心が高まっている。
 MOEは、計算化学や分子モデリング機能のほか、大量の化合物データを効率良く処理するケムインフォマティクス機能、たん白質の立体構造予測などのバイオインフォマティクス機能などを兼ね備えた統合ソフト。独自のプログラミング言語である“SVL”を中心にした開発環境が統合されており、アプリケーションを開発したりカスタマイズしたり、オリジナルのメニューやボタンバーを追加したりすることが簡単に行える。
MOEは、計算化学や分子モデリング機能のほか、大量の化合物データを効率良く処理するケムインフォマティクス機能、たん白質の立体構造予測などのバイオインフォマティクス機能などを兼ね備えた統合ソフト。独自のプログラミング言語である“SVL”を中心にした開発環境が統合されており、アプリケーションを開発したりカスタマイズしたり、オリジナルのメニューやボタンバーを追加したりすることが簡単に行える。
菱化システムでも、薬物と受容体のドッキングツール「ASEDock」や遺伝的アルゴリズムを用いたQSARツール「QuaSAR-Evolution」など、多数のSVLプログラム集を開発・公開している。これらは「SVLエクスチェンジ」(http://svl.chemcomp.com)として世界で100種類以上が登録されており、MOEの使用権を持っていればすべて無償で利用できることが特徴となっている。
今回の「MOE-AutoGPA」もそのプログラム集の1つとして提供されるもの。3D-QSAR法は、3次元構造に基づく特性を使用して、その特性と活性との相関をモデル化する方法で、活性分子の結合ポーズから周辺の空間的なポテンシャルを利用してモデルを構築するCoMFA法に代表される。ただ、活性分子の結合ポーズを得るために、構造を重ね合わせる方法の選択や、さまざまな重ね合わせの候補の中から最適なものを絞り込むことが難しかった。
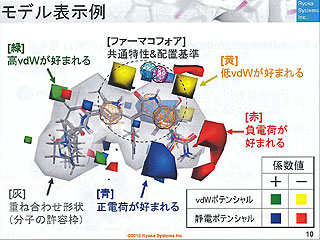
そこで、「MOE-AutoGPA」では、MOEに搭載されているファーマコフォアエルシデーション機能を利用してファーマコフォアベースの重ね合わせ(母核が共通していなくても重ね合わせることが可能)を実行。次に、すべての重ね合わせに対して、分子周辺に配置したグリッド上でのファンデルワールスおよび静電ポテンシャルを記述子としたQSARモデルを組み立てていく。最後に、すべての重ね合わせから得たモデルを予測性の指標を使ってランク付けし、最良モデルを選択する−という一連の手順を自動化した。
東海大学の平山令明教授の指導を得て開発したが、重ね合わせの基準となる構造が未知の場合でも、多数の重ね合わせからモデル構築し、最良モデルを選抜できる利点があるという。
また、ファーマコフォアおよび活性に関わるポテンシャルを示し、結合ポケットの形状と特性を把握しやすくするグラフィック表示機能を搭載。さらに、ファーマコフォアベースの3D-QSARスクリーニングツール「GPAScreen」、付加した置換基に対して予測活性値の高い二面角を提案する「GPADesign」といった周辺プログラムも揃っており、実用性も高い。