 今回の実用実験は、TSUBAMEの共用促進事業の産業利用トライアルユース(無償利用)における新規利用拡大枠として採択されたもの。これまでにTSUBAMEで利用実績がないアプリケーション分野であるとして利用が認められた。
今回の実用実験は、TSUBAMEの共用促進事業の産業利用トライアルユース(無償利用)における新規利用拡大枠として採択されたもの。これまでにTSUBAMEで利用実績がないアプリケーション分野であるとして利用が認められた。
ヒューリンクスがTSUBAMEでバーチャルスクリーニング実用実験
最大2,400コアの高並列環境を使用、40倍以上の高速化
2013.10.09−ヒューリンクスは、東京工業大学のスーパーコンピューター「TSUBAME」の共用促進事業を利用し、約111万化合物の大規模化合物ライブラリーからのバーチャルスクリーニングを実用実験として実施した。同社が販売権を持っている英クレセット社のスクリーニングソフト「Cresset blaze」を使い、大規模並列処理の有用性を探った。最大で「TSUBAME2.0」の200ノード/2,400コアを使用したが、貴重な経験が得られたという。
 今回の実用実験は、TSUBAMEの共用促進事業の産業利用トライアルユース(無償利用)における新規利用拡大枠として採択されたもの。これまでにTSUBAMEで利用実績がないアプリケーション分野であるとして利用が認められた。
今回の実用実験は、TSUBAMEの共用促進事業の産業利用トライアルユース(無償利用)における新規利用拡大枠として採択されたもの。これまでにTSUBAMEで利用実績がないアプリケーション分野であるとして利用が認められた。
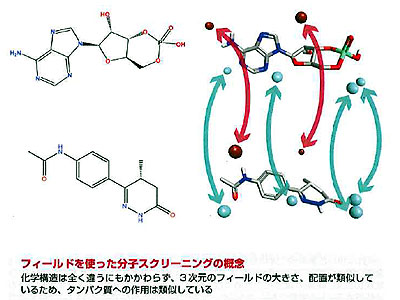
具体的には、英クレセットのバーチャルスクリーニングソフト「Cresset blaze」(旧製品名・FieldScreen)をTSUBAMEの並列処理環境で動かした。これは、クレセット独自の“フィールド”(分子の静電ポテンシャル・形状・疎水性などに基づく指標)情報を用いて、ライブラリー内のすべての化合物のフィールド情報と照合し、スコアリングして、一致度の高い化合物を探し出すソフト。フィールドに対する三次元の座標、強度の情報を総当たりで比較し、結果を並べ替えるため、ライブラリーが大きくなったり、分子が複雑になってフィールドポイントが増えたりすると、計算資源を多く消費することになる。
TSUBAMEは処理能力の大半をGPUで稼いでいるマシンだが、「blaze」はGPUに未対応であるため、CPUノードだけを用いて計算を行った。ただ、今回のトライアルユースの期間内には間に合わなかったが、GPUへの移植作業も並行して進めていったということだ。
実際の計算についてだが、まず専用に固定したノードに約111万件の化合物ライブラリーをリレーショナルデータベース(MySQLを使用)として設置。計算に使用するコア数に応じてデータベースを分割し、スクリーニングを実施して、その結果をウェブインターフェースを通して取得した。検索に用いたリガンドは非ヌクレオシド系逆転写酵素阻害薬の「Efavirenz」。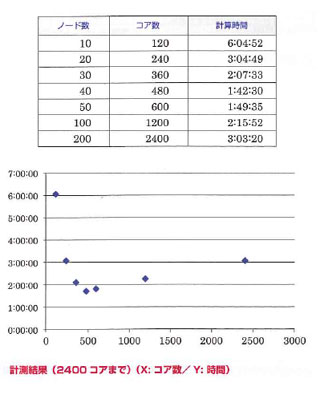
結論としては、40ノード/480コアの時が最もパフォーマンスが出ており、1時間42分で処理が終わった。1ノード/12コアでの理論上の計算時間は60時間であり、最大で40倍以上という並列化の効果が確認できたという。逆に、ノード数/コア数を増やしていくと、50ノード以上ではパフォーマンスが低下し、最大構成の200ノード/2,400コアでは3時間3分という結果になった。これは、処理の分割、処理の結合とそのソートにともなう通信のオーバーヘッドが徐々に増してしまったものだと考えられている。
いずれにしても、今後は一般的な傾向として、大規模並列環境での計算ニーズが増えると考えられることから、今回のTSUBAMEトライアルユースを通して実地での貴重な経験を積むことができたとしている。
******
<関連リンク>:
ヒューリンクス(トップページ)
http://www.hulinks.co.jp/
ヒューリンクス(クレセット製品紹介ページ)
http://www.hulinks.co.jp/software/cresset/index.html
東京工業大学(学術国際情報センター)
http://www.gsic.titech.ac.jp/