京都大学・ブラウン講師らが予測モデル構築の新手法
機械学習で化合物とタンパク質の結合性評価、少ないデータで高い予測精度
2017.03.24−京都大学医学研究科のJ.B.Brown(ジョン・ブラウン)講師と、チューリッヒ工科大学およびマサチューセッツ工科大学の研究グループは、医薬品分子と標的タンパク質との結合性を評価するための新しいモデル構築法を開発した。機械学習を効果的に用いることにより、大量データの中からモデル構築に重要な情報だけを効率的に選び出すことに成功。少ないデータで既存のモデルを上回る予測精度を達成したことで、創薬研究のコストダウンに役立つと期待されるという。3月6日、英フューチャーサイエンスの学術誌「Future Medicinal Chemistry」に論文「Active learning for computational chemogenomics」が掲載された。
 最近、創薬研究の世界でも人工知能(AI)が関心を集め、ビッグデータを活用したディープラーニングなどの手法による研究が熱を帯びている。しかし、ディープラーニングを用いることでしか得られない画期的な成果はまだ出ておらず、予測精度をわずかに上げるために膨大なデータが要求されるという課題がある。ブラウン講師らのグループでは、タンパク質と化合物との生理的な活性が発現する仕組みに明確な理論がない現状でいたずらにディープラーニングを実施しても、予測精度を飛躍的に向上させるような学習はそもそも難しいと判断。また、予測精度が上がったとしても、ディープラーニングではその理屈がわからないため、人の命に関わる医薬品開発に結びつけることには疑問があると指摘している。
最近、創薬研究の世界でも人工知能(AI)が関心を集め、ビッグデータを活用したディープラーニングなどの手法による研究が熱を帯びている。しかし、ディープラーニングを用いることでしか得られない画期的な成果はまだ出ておらず、予測精度をわずかに上げるために膨大なデータが要求されるという課題がある。ブラウン講師らのグループでは、タンパク質と化合物との生理的な活性が発現する仕組みに明確な理論がない現状でいたずらにディープラーニングを実施しても、予測精度を飛躍的に向上させるような学習はそもそも難しいと判断。また、予測精度が上がったとしても、ディープラーニングではその理屈がわからないため、人の命に関わる医薬品開発に結びつけることには疑問があると指摘している。
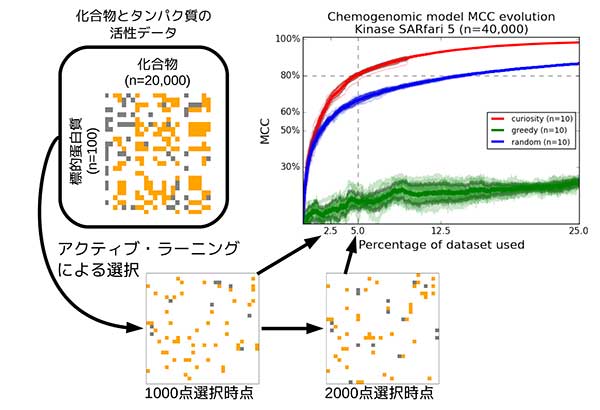
そこで今回の研究では、まずアクティブラーニングと呼ばれる機械学習手法を用い、タンパク質と化合物が結合する組み合わせと結合しない組み合わせを選び、それぞれの特徴を学習させた。学習した特徴から反応が起こるかどうかを予測し、予測結果を評価した上で精緻化に必要なデータをモデルに追加していった。
化合物とタンパク質との結合性を評価する予測モデルについては、決定木と呼ばれる解析手法を利用。決定木を500本つくり、解析結果の評価の仕方が異なる2種類のモデル(モデル名はCuriosityとGreedy)を用意した。そして実際の予測として、Gタンパク質共役型受容体(GPCR)とキナーゼ(酵素)の計3つのデータベースを使ってテストを実施。その結果、別図に示したように、ランダムに選んだデータからの予測に比べて、飛躍的に予測精度が高くなることがわかった。とくに、Curiosityモデルが優れた性能を示したが、すべての実験データの中の10〜20%を利用しただけで高い予測精度を達成できたことが大きな成果になるという。
ブラウン講師らは、どの化合物とタンパク質が予測精度を導くかを特定することにより、新薬候補化合物の探索が効率化されると結論している。また、今回の研究で予測に用いたデータベースには4万件ほどの化合物が含まれていたが、数百万単位のデータを使っても同様の予測精度を発揮できるかどうかを検証していきたいという。
******
<関連リンク>:
京都大学(ブラウン講師のホームページ)
http://statlsi.med.kyoto-u.ac.jp/~jbbrown/
京都大学(ジョン・ブラウン講師の教育研究活動データベース)
https://kyouindb.iimc.kyoto-u.ac.jp/j/mV4mL

ニュースファイルのトップに戻る
 最近、創薬研究の世界でも人工知能(AI)が関心を集め、ビッグデータを活用したディープラーニングなどの手法による研究が熱を帯びている。しかし、ディープラーニングを用いることでしか得られない画期的な成果はまだ出ておらず、予測精度をわずかに上げるために膨大なデータが要求されるという課題がある。ブラウン講師らのグループでは、タンパク質と化合物との生理的な活性が発現する仕組みに明確な理論がない現状でいたずらにディープラーニングを実施しても、予測精度を飛躍的に向上させるような学習はそもそも難しいと判断。また、予測精度が上がったとしても、ディープラーニングではその理屈がわからないため、人の命に関わる医薬品開発に結びつけることには疑問があると指摘している。
最近、創薬研究の世界でも人工知能(AI)が関心を集め、ビッグデータを活用したディープラーニングなどの手法による研究が熱を帯びている。しかし、ディープラーニングを用いることでしか得られない画期的な成果はまだ出ておらず、予測精度をわずかに上げるために膨大なデータが要求されるという課題がある。ブラウン講師らのグループでは、タンパク質と化合物との生理的な活性が発現する仕組みに明確な理論がない現状でいたずらにディープラーニングを実施しても、予測精度を飛躍的に向上させるような学習はそもそも難しいと判断。また、予測精度が上がったとしても、ディープラーニングではその理屈がわからないため、人の命に関わる医薬品開発に結びつけることには疑問があると指摘している。